Les posteurs les plus actifs de la semaine
| Aucun utilisateur |
Sujets les plus vus
Test post-hoc de Nemenyi
3 participants
Page 1 sur 1
 Test post-hoc de Nemenyi
Test post-hoc de Nemenyi
Bonjour à tous,
dans le cadre d'une étude de l'abondance des espèces en fonction d'un facteur qualitatif "Année" sur différents sites (on cherche à voir la recolonisation des espèces sur différents sites après travaux à différentes dates : voir si on retrouve un pattern dans les espèces présentes sur tous les sites en fonction du temps passé depuis la perturbation), j'ai effectué un test de kruskall wallis (Conditions d'application non respectées loi normale et homocédasticité)
Le test de kruskall wallis donne une pvalue largement inférieur à 0,05 ce qui me montre que le recouvrement de cette espèce varient bien en fonction de l'année. J'effectue ensuite un test post-hoc de nemenyi, et celui ci, lorsqu'il compare les années 2 à 2 ne donne pas de pvalue significative. Comment cela se fait-il ?
> kruskal.test(Juncus.effusus~Date_effacement,Totaux)
Kruskal-Wallis rank sum test
data: Juncus.effusus by Date_effacement
Kruskal-Wallis chi-squared = 19.728, df = 8, p-value = 0.01142
> posthoc.kruskal.nemenyi.test(Totaux$Juncus.effusus,Totaux$Date_effacement)
Pairwise comparisons using Tukey and Kramer (Nemenyi) test
with Tukey-Dist approximation for independent samples
data: Totaux$Juncus.effusus and Totaux$Date_effacement
2004 2009 2011 2012 2013 2014 2016 2017
2009 0.94 - - - - - - -
2011 0.92 1.00 - - - - - -
2012 0.99 1.00 1.00 - - - - -
2013 0.96 0.28 0.23 0.42 - - - -
2014 1.00 0.41 0.32 0.58 1.00 - - -
2016 1.00 0.73 0.66 0.89 0.98 1.00 - -
2017 0.99 0.25 0.17 0.35 1.00 1.00 1.00 -
2018 1.00 0.60 0.53 0.78 1.00 1.00 1.00 1.00
P value adjustment method: none
Warning message:
In posthoc.kruskal.nemenyi.test.default(Totaux$Juncus.effusus, Totaux$Date_effacement) :
Ties are present, p-values are not corrected.
Merci à vous
dans le cadre d'une étude de l'abondance des espèces en fonction d'un facteur qualitatif "Année" sur différents sites (on cherche à voir la recolonisation des espèces sur différents sites après travaux à différentes dates : voir si on retrouve un pattern dans les espèces présentes sur tous les sites en fonction du temps passé depuis la perturbation), j'ai effectué un test de kruskall wallis (Conditions d'application non respectées loi normale et homocédasticité)
Le test de kruskall wallis donne une pvalue largement inférieur à 0,05 ce qui me montre que le recouvrement de cette espèce varient bien en fonction de l'année. J'effectue ensuite un test post-hoc de nemenyi, et celui ci, lorsqu'il compare les années 2 à 2 ne donne pas de pvalue significative. Comment cela se fait-il ?
> kruskal.test(Juncus.effusus~Date_effacement,Totaux)
Kruskal-Wallis rank sum test
data: Juncus.effusus by Date_effacement
Kruskal-Wallis chi-squared = 19.728, df = 8, p-value = 0.01142
> posthoc.kruskal.nemenyi.test(Totaux$Juncus.effusus,Totaux$Date_effacement)
Pairwise comparisons using Tukey and Kramer (Nemenyi) test
with Tukey-Dist approximation for independent samples
data: Totaux$Juncus.effusus and Totaux$Date_effacement
2004 2009 2011 2012 2013 2014 2016 2017
2009 0.94 - - - - - - -
2011 0.92 1.00 - - - - - -
2012 0.99 1.00 1.00 - - - - -
2013 0.96 0.28 0.23 0.42 - - - -
2014 1.00 0.41 0.32 0.58 1.00 - - -
2016 1.00 0.73 0.66 0.89 0.98 1.00 - -
2017 0.99 0.25 0.17 0.35 1.00 1.00 1.00 -
2018 1.00 0.60 0.53 0.78 1.00 1.00 1.00 1.00
P value adjustment method: none
Warning message:
In posthoc.kruskal.nemenyi.test.default(Totaux$Juncus.effusus, Totaux$Date_effacement) :
Ties are present, p-values are not corrected.
Merci à vous
RoxanneF12- Nombre de messages : 6
Date d'inscription : 17/08/2019
 Re: Test post-hoc de Nemenyi
Re: Test post-hoc de Nemenyi
Manifestement, tu utilises R. L'implantation du posthoc dans R permet de choisir entre deux méthodes de calcul des p-values, celle par défaut étant basée sur une approche de type Tukey, l'autre sur une approche de type chi2. D'après l'aide, seule cette dernière option permet de corriger l'effet des 'tie' (ex-æquo en français). Le message te signale que tes données en comporte, ce qui peut expliquer ton problème.
Es-tu bien sûr que tu ne peux pas utiliser une Anova car :
1) quand on parle de normalité des données, il faut en fait comprendre celle des résidus, ce qui se teste a posteriori, une fois l'analyse faite et il existe des outils pour cela ;
2) l'Anova est relativement robuste et, éventuellement, on peut essayer d'utiliser une approche par moindres carrés pondérés pour corriger de l'hétéroscédasticité (utilisation de la fonction glm ou gls, voir le livre de Faraday accessible en ligne sur le site officiel de R, p62 et sq.)
Es-tu bien sûr que tu ne peux pas utiliser une Anova car :
1) quand on parle de normalité des données, il faut en fait comprendre celle des résidus, ce qui se teste a posteriori, une fois l'analyse faite et il existe des outils pour cela ;
2) l'Anova est relativement robuste et, éventuellement, on peut essayer d'utiliser une approche par moindres carrés pondérés pour corriger de l'hétéroscédasticité (utilisation de la fonction glm ou gls, voir le livre de Faraday accessible en ligne sur le site officiel de R, p62 et sq.)
Florent Aubry- Nombre de messages : 251
Date d'inscription : 02/11/2015
 Re: Test post-hoc de Nemenyi
Re: Test post-hoc de Nemenyi
Merci pour l'info. Pour l'ANOVA, voici mes lignes de code:
an<-lm(Persicaria.hydropiper~Date_effacement,Totaux)
x11();par(mfrow=c(2,2));plot(an)
dwtest(an)
leveneTest(an)
shapiro.test(an$res)
Totaux$Date_effacement<-as.factor(Totaux$Date_effacement)
TukeyHSD(aov(Persicaria.hydropiper~Date_effacement,Totaux))
J'effectue Shapiro pour la normalité, Levene et dwtest
Par contre, je ne sais pas comment analyser ces résultats concernant les conditions d'application. Peux-tu me donner quelques pistes pour savoir lire ces graphs et savoir si l'ANOVA est faisable ou non ? merci beaucoup !!
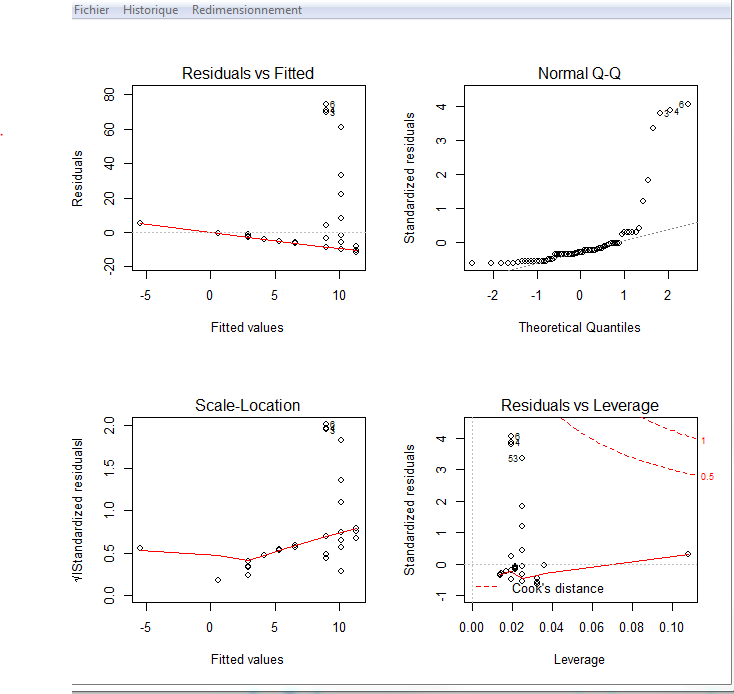
an<-lm(Persicaria.hydropiper~Date_effacement,Totaux)
x11();par(mfrow=c(2,2));plot(an)
dwtest(an)
leveneTest(an)
shapiro.test(an$res)
Totaux$Date_effacement<-as.factor(Totaux$Date_effacement)
TukeyHSD(aov(Persicaria.hydropiper~Date_effacement,Totaux))
J'effectue Shapiro pour la normalité, Levene et dwtest
Par contre, je ne sais pas comment analyser ces résultats concernant les conditions d'application. Peux-tu me donner quelques pistes pour savoir lire ces graphs et savoir si l'ANOVA est faisable ou non ? merci beaucoup !!

RoxanneF12- Nombre de messages : 6
Date d'inscription : 17/08/2019
 Re: Test post-hoc de Nemenyi
Re: Test post-hoc de Nemenyi
Le graphe le plus important est celui en haut à droite (Normal Q-Q plot). Dans ce graphe, les ordonnées représentent une description des résidus du modèle une fois ajusté, et en abscisses ces résidus si ils suivaient une loi normale. Idéalement, donc, tous les points devraient être sur la ligne pointillée, ce qui est loin d'être le cas ici. Le graphique indique même les numéros des observations qui s'éloignent vraiment de la normalité.
HTH, Eric.
HTH, Eric.

Eric Wajnberg- Nombre de messages : 1238
Date d'inscription : 14/09/2012
 Re: Test post-hoc de Nemenyi
Re: Test post-hoc de Nemenyi
Comme l'a écrit Eric, le graphe le plus important est le QQ-Plot. Il montre qu'il y a quelques points sont les résidus sont très importants et une zone de transition entre ces deux groupes de résidus. Le graphe du haut à gauche semble indiquer que le problème provient des groupes dont les valeurs estimées sont 10 et 9. Regarde de plus près ces groupes et regarde aussi de plus près les individus dont les numéros sont indiqués sur les graphiques. Vérifie s'ils sont cohérents avec tes critères d'inclusion (définition de la population testée) ou s'il n'y a pas eu de problème lors du recueil des données (mesure, transcription...) En cas de problème, tu peux avoir un justificatif pour les exclure de l'analyse, sinon pourquoi ne pas essayer un moindre carré pondéré (glm ou, préférentiellement, nlme::gls).
Quelques remarques sur ton script :
1) Totaux$Date_effacement<-as.factor(Totaux$Date_effacement) à faire avant l'analyse ;
2) inutile d'utiliser la fonction aov, passe plutôt par emmeans pour les tests post-hoc.
Quelques remarques sur ton script :
1) Totaux$Date_effacement<-as.factor(Totaux$Date_effacement) à faire avant l'analyse ;
2) inutile d'utiliser la fonction aov, passe plutôt par emmeans pour les tests post-hoc.
Florent Aubry- Nombre de messages : 251
Date d'inscription : 02/11/2015
 Sujets similaires
Sujets similaires» Test post-hoc
» le test post Hoc S-N-K
» Rsultats Post-hoc test
» test post-hoc non parametrique?
» Test Post-Hoc non paramétrique
» le test post Hoc S-N-K
» Rsultats Post-hoc test
» test post-hoc non parametrique?
» Test Post-Hoc non paramétrique
Page 1 sur 1
Permission de ce forum:
Vous ne pouvez pas répondre aux sujets dans ce forum