Les posteurs les plus actifs de la semaine
| Aucun utilisateur |
Sujets les plus vus
présentation des résultats de tests de Wilcoxon
2 participants
Page 1 sur 1
 présentation des résultats de tests de Wilcoxon
présentation des résultats de tests de Wilcoxon
Bonjour,
J'ai réalisé un série de test de Wilcoxon sous R.
Je voudrais savoir si ce type de présentation est correcte
(j'en ai toute une batterie, alors avant de trop fignoler la présentation pour chaque...):
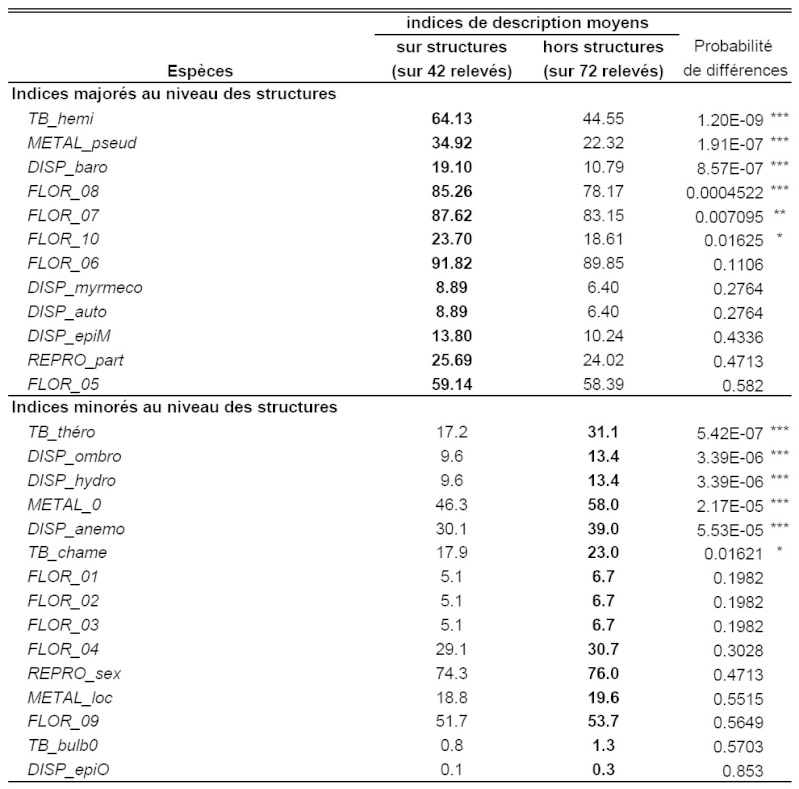
Je ne sais pas si cela est juste de mettre les effectifs mais d'omettre les valeurs des test W.
En plus, il y a le soucis des "indices de description moyens". Ne devrais-je pas plutôt mettre "les indices de description médians" étant en présence d'un test de Wilcoxon-Mann&Withney (donc non-paramétrique).
Dans ce cas, le dernier indice sur lequel porte le test "DISP_epiO" présenterait des "indices de description médians" sur et hors structure de 0 et 0.
Dois-je à votre avis dans ce cas créer un troisième groupe intitulé: "Indices indifférent à la présence de structures"?
Merci d'avance pour toute aide,
Tino
J'ai réalisé un série de test de Wilcoxon sous R.
Je voudrais savoir si ce type de présentation est correcte
(j'en ai toute une batterie, alors avant de trop fignoler la présentation pour chaque...):

Je ne sais pas si cela est juste de mettre les effectifs mais d'omettre les valeurs des test W.
En plus, il y a le soucis des "indices de description moyens". Ne devrais-je pas plutôt mettre "les indices de description médians" étant en présence d'un test de Wilcoxon-Mann&Withney (donc non-paramétrique).
Dans ce cas, le dernier indice sur lequel porte le test "DISP_epiO" présenterait des "indices de description médians" sur et hors structure de 0 et 0.
Dois-je à votre avis dans ce cas créer un troisième groupe intitulé: "Indices indifférent à la présence de structures"?
Merci d'avance pour toute aide,
Tino
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
???
personne n'a une idée?
Quelqu'un n'a jamais été amené à présenter ce genre de résultats?
Dans l'article suivant: http://www.isu.edu/bios/CV_Pub/Bowyer/CV_pdfs/62.pdf, ce sont bien les moyennes qui sont présentées... mais, est-ce juste?
PS: Sinon, j'ai bien sur oublié les erreurs standards associées aux moyennes... mais si ce sont les médianes que je dois présenter, est-ce qu'il y a un équivalent? Genre:
http://archive.numdam.org/ARCHIVE/RSA/RSA_1987__35_1/RSA_1987__35_1_5_0/RSA_1987__35_1_5_0.pdf
personne n'a une idée?
Quelqu'un n'a jamais été amené à présenter ce genre de résultats?
Dans l'article suivant: http://www.isu.edu/bios/CV_Pub/Bowyer/CV_pdfs/62.pdf, ce sont bien les moyennes qui sont présentées... mais, est-ce juste?
PS: Sinon, j'ai bien sur oublié les erreurs standards associées aux moyennes... mais si ce sont les médianes que je dois présenter, est-ce qu'il y a un équivalent? Genre:
http://archive.numdam.org/ARCHIVE/RSA/RSA_1987__35_1/RSA_1987__35_1_5_0/RSA_1987__35_1_5_0.pdf
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Bonsoir c'est quoi ce que tu appelles les indices de description moyen ?
Pour présenter un test de wilcoxon, il faut les n des deux distributions comparées, la valeur de la statistiques de wilcoxon associée et une p-value. et puis c'est tout.
Par contre ton intitulé "probabilité de différence" est faux
Pour présenter un test de wilcoxon, il faut les n des deux distributions comparées, la valeur de la statistiques de wilcoxon associée et une p-value. et puis c'est tout.
Par contre ton intitulé "probabilité de différence" est faux
Nik- Nombre de messages : 1606
Date d'inscription : 23/05/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
J'ai plusieurs types d'indices, ceux affichés dans ce tableau sont les indices "de description" de la végétation.Bonsoir c'est quoi ce que tu appelles les indices de description moyen ?
Etant donné que je compare ces indices entre n1 et n2 relevés, j'ai mis cet intitulé "indices de description moyen".
Pour des recouvrements de plantes en %, je mets "recouvrement moyen"...sur structure...et hors structure.
Mais dois-je mettre plutôt le recouvrement médian?
ok, dans le cas d'un test unique, mais dans le cas d'un tableau final? Il faut bien mettre la valeur moyenne (ou médiane) du facteur pour chaque modalité afin de "montrer" le sens de la différence. Dans ce cas, n'est-ce pas trop alourdir de mettre en plus la valeur du test dans le dit-tableau. J'ai un autre exemple d'article pour d'autres tests (fisher-exact) présentés de la même manière où la valeur du test n'est pas écrite (dans la même bonne revue que l'exemple précédent: Ecology, qui aurait laissé passé 2 fois la même erreur?).Pour présenter un test de wilcoxon, il faut les n des deux
distributions comparées, la valeur de la statistiques de wilcoxon
associée et une p-value. et puis c'est tout.
si je comprend bien, tu mettrais p-value ou probabilité?Par contre ton intitulé "probabilité de différence" est faux
C'est bien la probabilité que l'écart observé entre les deux jeux de données soit du au hasard?
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
ok...tant que tu expliques bien ton tableau dans une légende associée, pas de soucis. Pour ce qui est du recouvrement médian, peu importe car de toute façon la statistique du test que tu fais est sur les rangs donc que tu mettes la moyenne ou la médiane n'a pas plus de sens par rapport au test...C'est comme tu veux.J'ai plusieurs types d'indices, ceux affichés dans ce tableau sont les indices "de description" de la végétation.
Etant donné que je compare ces indices entre n1 et n2 relevés, j'ai mis cet intitulé "indices de description moyen".
Pour des recouvrements de plantes en %, je mets "recouvrement moyen"...sur structure...et hors structure.
Mais dois-je mettre plutôt le recouvrement médian?
Ce n'est pas une erreur, c'est un manqueJ'ai un autre exemple d'article pour d'autres tests (fisher-exact) présentés de la même manière où la valeur du test n'est pas écrite (dans la même bonne revue que l'exemple précédent: Ecology, qui aurait laissé passé 2 fois la même erreur?)
En tant qu'écologiste, on a souvent tendance à croire que la p-value se suffit à elle même mais c'est loin d'être le cas. Dans le cas des test non paramétriques, le pb est qu'on ne peut en plus pas juger de la différence car celle qui est mesurée est celle sur les rangs. On perd en grande partie l'information quantitative qui intéresse les biologistes. Il vaut mieux se tourner vers les tests de permutations qui sont bien plus puissants permettent des interprétations biologiques plus intéressante que simplement certifier que la différence existe.
la p-value est la proba de rejeter l'hypothèse H0 alors qu'elle est vraie.si je comprend bien, tu mettrais p-value ou probabilité?
C'est bien la probabilité que l'écart observé entre les deux jeux de données soit du au hasard?
Donc la proba de différence c'est 1 - p-value.
Nik- Nombre de messages : 1606
Date d'inscription : 23/05/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Bonjour,
Dans le cas du test de wilcoxon et surtout dans un cas de comparaisons multiples, je ne chargerais pas l'info avec la valeur du test qui dans le cas de tests non paramétriques sont très difficiles à interpréter. Faire attention : la statistique du test ne te donne pas le sens de la différence et l'hypothèse alternative n'est pas que la somme des rangs d'un des deux groupes est inférieure ou supérieure à la somme des rangs du second groupe.
Perso je mettrai la moyenne plutôt que la médiane dans le tableau qui est pour beaucoup de gens beaucoup plus explicite (en rajoutant l'écart type) même si elle ne veut pas dire grand chose quand ta distribution est fortement asymétrique. De plus le test de wilcoxon est considéré par beaucoup comme l'alternative non paramétrique au test de student.
Pourquoi être parti sur le test de wilcoxon ? Problème de variance non égale entre les groupes ou des distributions qui ne suivent pas une loi normale ? Comme l'a dit Nik quand on peut rester en quantitatif c'est quand même mieux.
Je simpliderai aussi la représentation des p-values avec un truc du genre : >0.05 , <0.05, <0.01 et <0.001.
micros
Dans le cas du test de wilcoxon et surtout dans un cas de comparaisons multiples, je ne chargerais pas l'info avec la valeur du test qui dans le cas de tests non paramétriques sont très difficiles à interpréter. Faire attention : la statistique du test ne te donne pas le sens de la différence et l'hypothèse alternative n'est pas que la somme des rangs d'un des deux groupes est inférieure ou supérieure à la somme des rangs du second groupe.
Perso je mettrai la moyenne plutôt que la médiane dans le tableau qui est pour beaucoup de gens beaucoup plus explicite (en rajoutant l'écart type) même si elle ne veut pas dire grand chose quand ta distribution est fortement asymétrique. De plus le test de wilcoxon est considéré par beaucoup comme l'alternative non paramétrique au test de student.
Pourquoi être parti sur le test de wilcoxon ? Problème de variance non égale entre les groupes ou des distributions qui ne suivent pas une loi normale ? Comme l'a dit Nik quand on peut rester en quantitatif c'est quand même mieux.
Je simpliderai aussi la représentation des p-values avec un truc du genre : >0.05 , <0.05, <0.01 et <0.001.
micros
Invité- Invité
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Nik:
Nik:
je ne connais malheureusement pas toutes les ficelles des stats...
micros corpus:
micros corpus:
micros corpus:
Ok, grossière erreur de ma part!la p-value est la proba de rejeter l'hypothèse H0 alors qu'elle est vraie.
Donc la proba de différence c'est 1 - p-value.
Nik:
qu'entend-tu par tests de permutation: monte carlo?En tant qu'écologiste, on a souvent tendance à croire que la p-value se
suffit à elle même mais c'est loin d'être le cas. Dans le cas des test
non paramétriques, le pb est qu'on ne peut en plus pas juger de la
différence car celle qui est mesurée est celle sur les rangs. On perd
en grande partie l'information quantitative qui intéresse les
biologistes. Il vaut mieux se tourner vers les tests de permutations
qui sont bien plus puissants permettent des interprétations biologiques
plus intéressante que simplement certifier que la différence existe.
je ne connais malheureusement pas toutes les ficelles des stats...
micros corpus:
écart-type ou écart-type de la moyenne (erreur standard)?Perso je mettrai la moyenne plutôt que la médiane dans le tableau qui
est pour beaucoup de gens beaucoup plus explicite (en rajoutant l'écart
type)
micros corpus:
en fait, problème de beaucoup de tests... avec parfois peu d'individus, des distributions non normales... donc j'ai décidé de tout faire en non paramétrique afin de pouvoir tout mettre dans des tableaux uniques... je perd en puissance, certes, mais je considère que si un test non paramétrique met en évidence des différences, un test paramétrique l'aurait aussi fait... seules les différences faibles échappent à ces tests non paramétriquesPourquoi être parti sur le test de wilcoxon ? Problème de variance non
égale entre les groupes ou des distributions qui ne suivent pas une loi
normale ? Comme l'a dit Nik quand on peut rester en quantitatif c'est
quand même mieux.
micros corpus:
c'est ce que j'ai représenté par des étoiles... mais c'est sur, les valeurs pourraient au moins être arrondies...Je simpliderai aussi la représentation des p-values avec un truc du genre : >0.05 , <0.05, <0.01 et <0.001.
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
c'est ce que j'ai représenté par des étoiles... mais c'est sur, les valeurs pourraient au moins être arrondies...
alors les deux infos sont redondantes et une seule des deux est utile (les étoiles ou la valeur de p-value).
Un truc m'échappe, tu dis avoir parfois peu d'individus, mais si je regarde ton tableau on voit pour une colonne 42 relevés et pour l'autre 72 relevés. Tu n'utilises pas a chaque fois ces 114 relevés dans tes analyses ? Si ce n'est pas le cas alors tu devrais spécifier les effectifs de chaque classe à chaque fois.en fait, problème de beaucoup de tests... avec parfois peu d'individus,
des distributions non normales... donc j'ai décidé de tout faire en non
paramétrique afin de pouvoir tout mettre dans des tableaux uniques...
je perd en puissance, certes, mais je considère que si un test non
paramétrique met en évidence des différences, un test paramétrique
l'aurait aussi fait... seules les différences faibles échappent à ces
tests non paramétriques
A près si tu as pas mal de zéro ça peut mettre le bazard dans le calcul des tests et il te faut faire attention que ton test prenne bien en compte les exaequos.
Perso je mettrais aussi l'écart type.
micros
Invité- Invité
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Un test de permutation se base sur une statistique de test et on va évaluer la sgnificativité en faisant des tirages aléatoire dans les valeurs observées. Dans ton casqu'entend-tu par tests de permutation: monte carlo?
- 2 distributions A et B avec respectivement nA et nB valeurs.
- tu cacules la différence entre les deux moyennes observées : nAbar - nBbar = Mobs
- ensuite tu regroupes toutes tes données. tu tires aléatoirement nA valeurs, dont tu fais la moyenne et avec les nB autres valeurs tu fais aussi une moyenne. Tu fais la différence : nAbarSim - nBbarSim = Msim.
-Tu reproduis l'étape précédente autant de fois qu'il y de combinaison possible de nA tirage parmi nA+nB (si ça dépasse 1000 en général on s'arrête
Pour la p-value :
- la p-value unilatérale est calculée comme la proportion des valeurs simulées (Msim) supérieure ou égale à Mobs.
- la p-value bilatérale est calculée comme la proportion des valeurs simulées dont la valeur absolue est supérieure ou égale à la valeur absolue de Mobs.
voilà.
Nik- Nombre de messages : 1606
Date d'inscription : 23/05/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
micros corpus :
Après je découpe les 42 relevés sur structures en 2 classes (structures en creux, structures en élévation) et ainsi de suite... (élévation=mur+talus; creux=fosse+fossés)... et à chaque fois je réitère la série de test... donc à la fin il me reste peu d'individus dans chaque modalité.
Nik:
L'exemple de mon tableau est mal choisi: c'est mon test de base sur l'ensemble de mes relevés.Un truc m'échappe, tu dis avoir parfois peu d'individus, mais si je
regarde ton tableau on voit pour une colonne 42 relevés et pour l'autre
72 relevés. Tu n'utilises pas a chaque fois ces 114 relevés dans tes
analyses ? Si ce n'est pas le cas alors tu devrais spécifier les
effectifs de chaque classe à chaque fois.
Après je découpe les 42 relevés sur structures en 2 classes (structures en creux, structures en élévation) et ainsi de suite... (élévation=mur+talus; creux=fosse+fossés)... et à chaque fois je réitère la série de test... donc à la fin il me reste peu d'individus dans chaque modalité.
Nik:
Je comprend bien le fonctionnement, celà me semble en effet plus adapté à mes données et mes problématiques. Mais d'un point de vue pratique, j'ai jamais essayé. Je travaille sous R, il doit bien exister un package adéquat?Un test de permutation se base sur une statistique de test et on va
évaluer la sgnificativité en faisant des tirages aléatoire dans les
valeurs observées. Dans ton cas
- 2 distributions A et B avec respectivement nA et nB valeurs.
- tu cacules la différence entre les deux moyennes observées : nAbar - nBbar = Mobs
-
ensuite tu regroupes toutes tes données. tu tires aléatoirement nA
valeurs, dont tu fais la moyenne et avec les nB autres valeurs tu fais
aussi une moyenne. Tu fais la différence : nAbarSim - nBbarSim = Msim.
-Tu
reproduis l'étape précédente autant de fois qu'il y de combinaison
possible de nA tirage parmi nA+nB (si ça dépasse 1000 en général on
s'arrête) et voilà. Tu as une statistique robuste.
Pour la p-value :
- la p-value unilatérale est calculée comme la proportion des valeurs simulées (Msim) supérieure ou égale à Mobs.
-
la p-value bilatérale est calculée comme la proportion des valeurs
simulées dont la valeur absolue est supérieure ou égale à la valeur
absolue de Mobs.
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
bah...est il bien nécessaire de poser la question en ce sens ?Je travaille sous R, il doit bien exister un package adéquat?
regardes la fonction randtest ou as.randtest dans le package ade4...ça marche bien et il y a aussi la fonction plot.randtest qui donne le graphique à mettre dans la publi ou le rapport ... trop top
Il y a peut être d'autres packages et fonction je n'ai jamais regardé.
Nik
Nik- Nombre de messages : 1606
Date d'inscription : 23/05/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
j'ai pas trop compris le fonctionnement de cette fonction... du coup, j'ai écrit les codes suivants en fonction de ce que tu as énoncé précédemment:regardes la fonction randtest ou as.randtest dans le package ade4...
p_value_unilatérale:
- Code:
> table<-numeric(10000)
> for(i in 1:10000)
+ {var<-sample(indice,nrow(groix_fin_rec),replace=F)
+ temp<-data.frame(Structure,var)
+ temp1<-subset(temp,Structure=="sur_structure")
+ moy1<-mean(temp1$var)
+ temp2<-subset(temp,Structure=="hors_structure")
+ moy2<-mean(temp2$var)
+ table[i]<-moy1-moy2}
> data<-data.frame(groix_fin_rec)
> tempo1<-subset(data,Structure=="sur_structure")
> moy1<-mean(tempo1$indice)
> tempo2<-subset(data,Structure=="hors_structure")
> moy2<-mean(tempo2$indice)
> delta<-moy1-moy2
> tabletf<-drop(delta>table)
> tablet<-subset(tabletf,tabletf==TRUE)
> p_value<-ifelse(0>delta,length(tablet)/(10000+1),1-length(tablet)/(10000+1))
> p_value
p_value_bilatérale:
- Code:
> table<-numeric(10000)
> for(i in 1:10000)
+ {var<-sample(indice,nrow(groix_fin_rec),replace=F)
+ temp<-data.frame(Structure,var)
+ temp1<-subset(temp,Structure=="sur_structure")
+ moy1<-mean(temp1$var)
+ temp2<-subset(temp,Structure=="hors_structure")
+ moy2<-mean(temp2$var)
+ table[i]<-abs(moy1-moy2)}
> data<-data.frame(groix_fin_rec)
> tempo1<-subset(data,Structure=="sur_structure")
> moy1<-mean(tempo1$indice)
> tempo2<-subset(data,Structure=="hors_structure")
> moy2<-mean(tempo2$indice)
> delta<-abs(moy1-moy2)
> tabletf<-drop(delta>table)
> tablet<-subset(tabletf,tabletf==TRUE)
> p_value<-length(tablet)/(10000+1)
> p_value
j'espère que c'est juste...
Effectivement on gagne en puissance, d'après les essais que j'ai réalisé un p de 0.01016 en wilcoxon passe à 0.0028 pour des permutations en unilatéral et 0.0079 en bilatéral.
Par contre je ne sais pas quand utiliser la p-value unilatérale ou la p-value bilatérale.
Au fait, as-tu des références biblio pour ce test?
Merci d'avance,
Tino
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Il n'est pas évident que les tests de permutation soient plus adaptés dans le cas ou tu compares les groupes avec de très faibles effectifs. De plus ici tu ne peux pas comparer les différences de p-value que tu observes entre le test de wlicoxon et le code R, puisque dans un cas tu travailles sur la somme des rangds et dans l'autre sur la différence entre tes deux moyennes et donc en paramétrique, autrement dit les deux choses n'ont rien à voir. Si tu fais des permutations c'est pour recalculer la statistique de ton test pas pour calculer une statistique qui n'a rien à voir. Pour ce qui est d'utiliser le bilateral ou l'unilateral, ça dépend de ton hypothèse alternative : est-ce que les moyennes sont différentes (bilatérale), est-ce que la moyenne du groupe sur_structure est supérieure (ou inférieure) à la moyenne du groupe hors_structure (unilatérale).
micros
P.S: le code R pour reproduire ce qui a été fait ci dessus mais de façon un peu plus optimisée (essayer de ne pas utiliser des noms d'objets qui sont aussi des noms de fonction) :
micros
P.S: le code R pour reproduire ce qui a été fait ci dessus mais de façon un peu plus optimisée (essayer de ne pas utiliser des noms d'objets qui sont aussi des noms de fonction) :
- Code:
table <- replicate(10000,{
var<-sample(indice,nrow(groix_fin_rec),replace=F)
abs(diff(tapply(var,Structure,mean)))
})
# je ne pense pas que la valeur absolue soit adéquate non plus
Invité- Invité
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Je sais, mais je voulais juste dire que d'après les p-value de mes quelques tests, il y a de grandes chances de voir apparaître des différences (de moyennes) dans des cas où wilcoxon n'avais pas vu de différences de rangs.micros corpus a écrit:De plus ici tu ne peux pas comparer les différences de p-value que tu observes entre le test de wlicoxon et le code R, puisque dans un cas tu travailles sur la somme des rangds et dans l'autre sur la différence entre tes deux moyennes et donc en paramétrique, autrement dit les deux choses n'ont rien à voir.
qu'entends-tu pas là ?micros corpus a écrit:Si tu fais des permutations c'est pour recalculer la statistique de ton test pas pour calculer une statistique qui n'a rien à voir.
c'est bien le problème...micros corpus a écrit:Pour ce qui est d'utiliser le bilateral ou l'unilateral, ça dépend de ton hypothèse alternative : est-ce que les moyennes sont différentes (bilatérale), est-ce que la moyenne du groupe sur_structure est supérieure (ou inférieure) à la moyenne du groupe hors_structure (unilatérale).
faire 2 tests unilatérals (supérieur et inférieur) font apparaître des p-value inférieures à un test bilatéral...???
en général quand on teste des différences de moyennes, on veut en savoir le sens, alors quel est l'intérêt du bilatéral?
Grands mercis pour le code, effectivement, c'est plus rapide à l'exécution et celà donne les mêmes valeurs.micros corpus a écrit:
- Code:
table <- replicate(10000,{
var<-sample(indice,nrow(groix_fin_rec),replace=F)
abs(diff(tapply(var,Structure,mean)))
})
# je ne pense pas que la valeur absolue soit adéquate non plus
concernant la p-value finale, pense-tu qu'il faut utiliser 10000 ou 10000+1 comme j'ai fait?
Qu'entends-tu par "je ne pense pas que la valeur absolue soit adéquate non plus"?
Tino
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
tinomulot a écrit:qu'entends-tu pas là ?micros corpus a écrit:Si tu fais des
permutations c'est pour recalculer la statistique de ton test pas pour
calculer une statistique qui n'a rien à voir.
J'entends qu'ici tu compares des choses qui n'ont rien à voir. Comparer
la p-value de ton test de wilcoxon, et les p-values que tu obtiens sur les différences de moyenne ça n'a rien à voir ! Puisque tu ne fais pas la même chose, dans un cas tu regardes la somme des rangs et dans l'autre cas une différence de moyenne. De plus je trouve ça dangereux de comparer des moyennes de la sorte, dans chaque test de comparaison de moyenne paramétrique la variance est prise en compte ce qui n'est pas le cas ici. Si tu veux vraiment comparer ton test de wilcoxon avec un test sur les permutation alors tu permuttes tes données (N fois) tu récupères les statistiques de ces tests et tu compares si la statistique de ton test que tu obtiens sur les données brutes avec la distribution des statistiques obtenues par permutation.
Si je regarde plus haut, tu dis que la distribution n'est pas normale, alors dans ce cas la je pense que la moyenne n'a pas de sens.
Après l'histoire de choisir un test unlitateral ou biolateral ne dépend pas de la valeur de la p-value mais de tes hypothèses de travails ! Dans le cas d'un test bilateral tu n'as pas d'hypothèse concernant l'effet que ça pourrait avoir sur la différence de la moyenne, alors que si utilises un test unilatéral tu as une hypothèse.
Exemple : tu utilises deux type d'engrais pour produire du maïs et tu cherches a savoir si les productions sont différentes ou non selon le type d'engrais utuliser. Si tu ne connais pas les engrais tu vas juste chercher à les comparer, alors tu vas utiliser un test bilatéral.
Après si tu compares des productions de maïs dans des champs avec et sans engrais, tu as pour hypothèse que l'engrais augmente le rendement, alors ce que tu cherches à savoir c'est si la production est plus forte avec l'engrais que sans. La tu es dans le cas d'un test unliatéral.
J'espère que c'est plus clair pour toi.
moi je prendrais N=9999 permutations + 1 observation.
micros
Invité- Invité
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
merci pout tout ces conseils,
voila ma fonction finale,
j'ai notamment modifié "tabletf<-drop(table>delta)" en "tabletf<-drop(table>=delta)"
encore merci,
Tino
PS: dans le cas de valeurs manquantes NA pour la variable considérée (mais pas pour la modalité), pensez-vous qu'il faille éliminer les lignes correspondantes ou distribuer aléatoirement les NA? Je pense à la première solution afin d'avoir des moyennes aléatoires basées sur le même nombre d'individus que les moyennes observées, mais j'ai un doute...
voila ma fonction finale,
j'ai notamment modifié "tabletf<-drop(table>delta)" en "tabletf<-drop(table>=delta)"
- Code:
permutino<-function(fichier,variable,modalite)
{attach(fichier)
table <- replicate(9999,{
tempo<-sample(variable,nrow(fichier),replace=F)
abs(diff(tapply(tempo,modalite,mean)))
})
delta<-abs(diff(tapply(variable,modalite,mean)))
tabletf<-drop(table>=delta)
tablet<-subset(tabletf,tabletf==TRUE)
p_value<-length(tablet)/(9999+1)
nb_rel<-tapply(variable,modalite,length)
moyenne<-tapply(variable,modalite,mean)
err_std<-sqrt(tapply(variable,modalite,var)/tapply(variable,modalite,length))
result<-rbind(nb_rel, moyenne,err_std)
print(result)
print(cat("\n","p-value",p_value,"\n"))
detach(fichier)
}
encore merci,
Tino
PS: dans le cas de valeurs manquantes NA pour la variable considérée (mais pas pour la modalité), pensez-vous qu'il faille éliminer les lignes correspondantes ou distribuer aléatoirement les NA? Je pense à la première solution afin d'avoir des moyennes aléatoires basées sur le même nombre d'individus que les moyennes observées, mais j'ai un doute...
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Re,
J'ai trouvé un cours de biostatistique qui peut te servir comme référence pour le test de permutation que tu fais. Néanmoins, il y a une restriction très forte à l'utilisation de ce test qui est la suivante :
Il faut que les variances de tes deux échantillons soient équivalentes !
Si ce n'est pas le cas tu peux jeter à la poubelle les conclusions de tes tests.
http://homepages.ulb.ac.be/~aleveque/epitraumac/pdf-ppt/bootstrap2.pps
Je te laisse regarder la diapositive 25.
micros
J'ai trouvé un cours de biostatistique qui peut te servir comme référence pour le test de permutation que tu fais. Néanmoins, il y a une restriction très forte à l'utilisation de ce test qui est la suivante :
Il faut que les variances de tes deux échantillons soient équivalentes !
Si ce n'est pas le cas tu peux jeter à la poubelle les conclusions de tes tests.
http://homepages.ulb.ac.be/~aleveque/epitraumac/pdf-ppt/bootstrap2.pps
Je te laisse regarder la diapositive 25.
micros
Invité- Invité
 Re: présentation des résultats de tests de Wilcoxon
Re: présentation des résultats de tests de Wilcoxon
Re,
Merci pour l'info,
Tino
PS: Je teste l'égalité de variances par bootstrap?
Merci pour l'info,
Tino
PS: Je teste l'égalité de variances par bootstrap?
tinomulot- Nombre de messages : 17
Age : 44
Localisation : Neuvile (Correze)
Date d'inscription : 02/10/2008
 Sujets similaires
Sujets similaires» présentation des résultats Mann Whitney / Wilcoxon
» Resultats d'un test Wilcoxon Mann Whitney
» Test de Wilcoxon et tests des rangs signés
» Récupérer depuis R les résultats de tests sous forme de tabl
» Wilcoxon
» Resultats d'un test Wilcoxon Mann Whitney
» Test de Wilcoxon et tests des rangs signés
» Récupérer depuis R les résultats de tests sous forme de tabl
» Wilcoxon
Page 1 sur 1
Permission de ce forum:
Vous ne pouvez pas répondre aux sujets dans ce forum